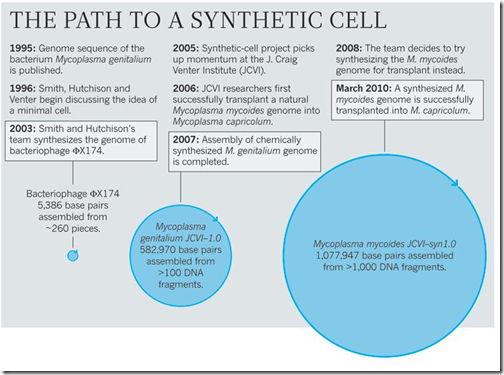
estamos em 2012. cientistas acabam de construir [escrevendo em software] e rodar [em um “cluster” de 128 processadores] uma simulação computacional “completa” do ciclo de vida de um organismo vivo. o objeto em estudo é tão simples que nem parece vivo: trata-se do mycoplasma genitalium, de meros 525 genes, que teve a honra de ser o primeiro organismo a ter seu DNA sequenciado [em 1995] e depois sintetizado do zero, em 2007.
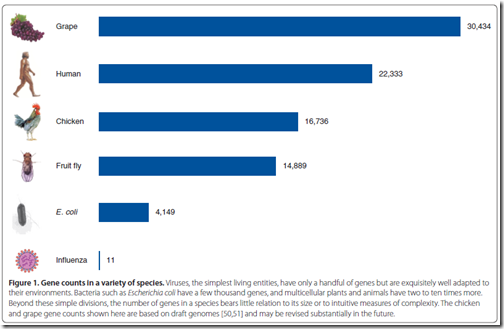
pelas contas do dia, eu e você temos 22.333 genes [mais ou menos, o número varia em ~80 genes por indivíduo…], que cabem em menos de 800 megabytes. o qualquer celular básico armazena a informação do seu DNA. cabe em um CD, fácil. nosso “código” é maior que o das galinhas, que têm 16.736 genes, mas menor do que o da uva comum, que tem 30.434 genes. aquela gripe “mortal” que a gente tem, vez por outra, é causada por um carinha que tem apenas 11 genes. pense.
o povo que escreveu o simulador do m. genitalium levou em conta mais de 900 artigos científicos e 1.900 experimentos para construir seu modelo. simular uma única divisão celular, neste caso, leva cerca de dez horas e gera 500 megabytes de dados. a célula real, em ambiente real, também leva dez horas pra completar uma divisão. se [ou quando] o tempo de simulação for centenas, milhares ou milhões de vezes mais rápido do que o real, vamos aprender coisas sobre m. genitalium, em semanas ou dias –em software, para as quais seriam necessários anos ou décadas em laboratório. vai ver a gente descobre, rápido, como a coisa causa artrite e muito mais. e como tratar isso. rápido.
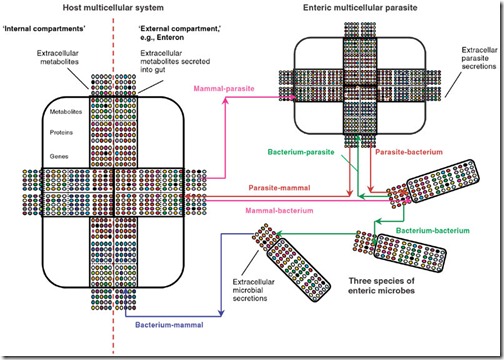
pra isso, vamos ter que juntar muito mais textos e experimentos, milhões, talvez, pra simular o organismo onde o parasita se aloja: nós. e, aí, simular a interação dos dois, nós e eles. simular humanos não vai ser 40 vezes mais caro do que simular uma bactéria de quinta categoria. a complexidade do problema não é linear, aqui. aliás, vai dar muito mais trabalho do que se pensa: o organismo humano somos “nós” e mais uns 500 tipos de bactérias, que juntas têm uns 100 trilhões de células, cerca de um quilo do nosso peso. nós, por acaso, “só” temos uns 10 trilhões de células. o que significa que eu e você carregamos um monte de aliens. essenciais pra nossa sobrevivência. pois não somos um organismo, mas uma federação deles.
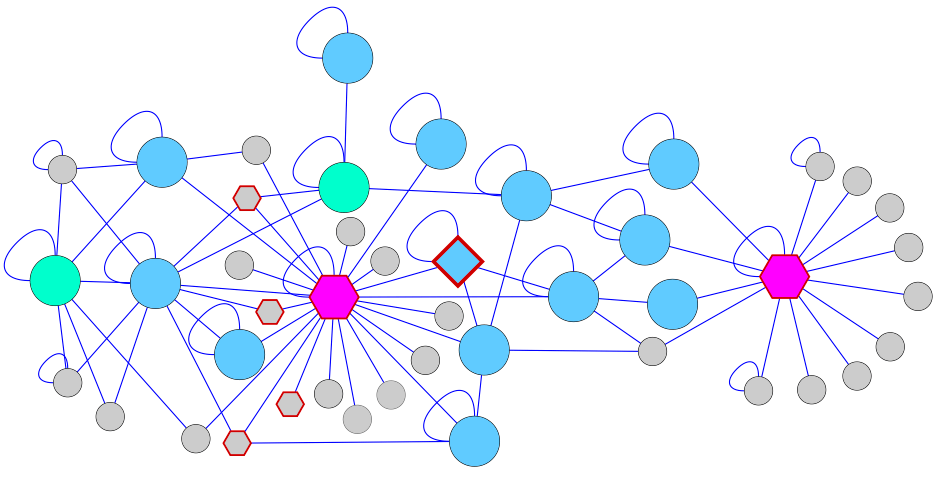
resultado? olha só parte das interações que acontecem no intestino e a quantidade de trocas possíveis quando “tomamos um remédio”, por exemplo…
chegar a um modelo de simulação razoável de uma bactéria de uma única célula foi simples, considerando o desafio de simular um humano, um superorganismo. o vídeo da “divisão” simulada, acelerado, está abaixo. dois minutos, só. veja.
entre descobrir o genoma do m. genitalium e fazer uma simulação completa, que nos explica muita coisa do bicho, foram 17 anos. o genoma humano foi impresso pela primeira vez em 2000, em versão beta, e foi dado por decodificado em 2006, 16 anos depois do início do projeto. se fosse apostar, eu diria que vamos ter uma boa simulação do superorganismo humano neste século. mas não tenha pressa: o maior supercomputador do planeta, no momento, é sequoia e, com 1.572.864 unidades de processamento, tem menos da metade da capacidade computacional que se imagina necessária para simular um único cérebro.
vamos chegar lá. vamos simular humanos. vamos simular populações humanas inteiras e suas interações. vamos simular muito mais que isso, talvez tudo. se você tiver paciência para ler o link anterior, é capaz de ficar desconfiado de que já estamos vivendo em uma simulação computacional… mas pode ser só imaginação. aliás, eu e você podemos ser só imaginação, simulação… quem sabe?…